González-Bello
Liñas de investigación
Antibacterianos: Aproximacións innovadoras para enfrontar o desafío das súper-bacterias. Dianas terapéuticas inexploradas. Fármacos con Novos Modos de Acción. Novos kits de diagnóstico.
Investigador(es) principal/principais
Membros do grupo
| Maneiro Rey, María |
Inv. Posdoutoral |
|
| Canabal Facón, Rafael |
Inv. Predoutoral |
|
| Casabella Amieiro, Braulio |
Inv. Predoutoral |
|
| Outeiral Valiño, María Carmen |
Inv. Predoutoral |
|
| Suárez de Cepeda Fuentes, Pilar |
Inv. Predoutoral |
Investigación
Estratexias para Desbloquear a Resistencia Bacteriana
A estratexia actual no descubrimento de fármacos para o tratamento das enfermidades infecciosas por parte da industria farmacéutica, non cambiou moito nos últimos 60 anos. Esta implica principalmente modificacións de medicamentos anteriores que inciden sobre un pequeno número de funcións bacterianas e do mesmo tipo, para facelos máis eficaces e resistentes contra as bacterias resistentes. Con todo, a resistencia a estes obxectivos biolóxicos é xeneralizada e ben coñecida. Por tanto, necesítase con urxencia unha nova estratexia no descubrimento de antibióticos para controlar o crecente desenvolvemento e a propagación das SÚPERBACTERIAS, que son patóxenos que mesmo se volveron resistentes a múltiples tipos de antibióticos. O noso grupo de investigación céntrase no desenvolvemento de novos axentes antibacterianos con mecanismos de acción INNOVADORES para tratar infeccións mortais causadas polos patóxenos Gram-negativos multirresistentes, en especial para o tratamento das infeccións causadas polas bacterias prioritarias recentemente identificadas pola OMS, é dicir, Acinetobacter baumannii, Pseudomonas aeruginosa e Enterobacteriaceae. Estes patóxenos son moi perigosos en pacientes cun sistema inmunitario débil sometidos a quimioterapia contra o cancro, cirurxía xeral, transplantes de órganos e diálise, entre outros, para quen nosa capacidade de tratar as infeccións secundarias é crucial. Estamos a estudar ademais, o potencial de dianas bacterianas inexploradas para o desenvolvemento de novos axentes contra a tuberculose, enfermidade considera unha prioridade de saúde mundial.
Actualmente no noso grupo de investigación explóranse tres estratexias principais que inciden sobre a VIABILIDADE BACTERIANA, a RESISTENCIA BACTERIANA e a COMUNICACIÓN ENTRE BACTERIAS.
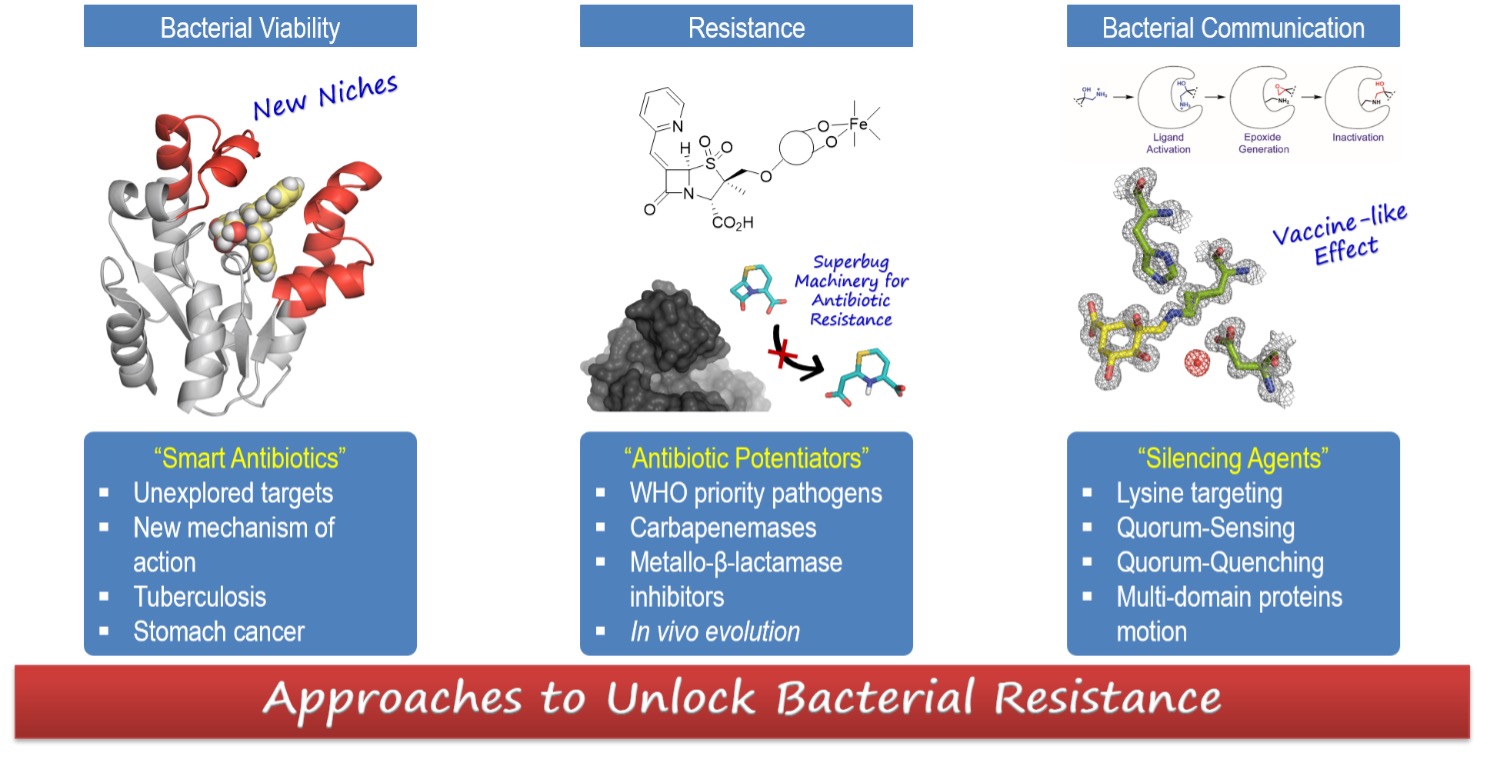
ANTIBIOTICOS INTELIXENTES – Estamos a estudar o potencial terapéutico de varios enzimas da ruta do ácido siquímico (siquimato quinasa, EPSP sintasa) para o descubrimento de novos antibióticos. Estas dianas terapéuticas pouco exploradas son esenciais para a viabilidade da bacteria. En base ao movemento esencial do enzima para a expulsión do produto do centro activo (deseño baseado no movemento), identificamos potentes inhibidores competitivos reversibles que estabilizan unha conformación aberta inactiva para a catálise (J. Med. Chem. 2016, 59, 5471, Chem. Eur. J. 2016, 22, 17988; Org. Chem. Front. 2019, 6, 2514). Tamén desenvolvemos inhibidores mediante a rigidificación da conformación activa de alta enerxía que recoñece o enzima para a catálise (J. Am. Chem. Soc. 2013, 135, 12366). Utilizamos un enfoque pro-fármaco para a internalización dos mesmos na bacteria.
POTENCIADORES DOS ANTIBIOTICOS – Estamos a traballar no desenvolvemento de novos antibióticos adxuvantes capaces de bloquear o mecanismo máis relevante da resistencia bacteriana: a inactivación enzimática do antibiótico (β-lactamas) mediante a acción das β-lactamasas. Desde o punto de vista do descubrimento de fármacos, esta terapia combinada (antibiótico + antibiótico adxuvante) ten a vantaxe de que non é necesario investir esforzos na tan custosa e complexa identificación de novas dianas terapéuticas. En concreto, centrámonos nas β-lactamasas mais perigosas, as carbapenemasas de clase D (OXA-23, OXA-24/40, OXA-48), as metalo-β-lactamasas, e as cromosómicas de tipo C en P. aeruginosa (AmpC). que son producidas por as súperbacterias más peligrosas no entorno hospitalario. Desenvolvemos diversas sulfonas baseadas en penicilina que forman aductos estables de indolizidina co enzima, permitindo recuperar, tanto in vitro como in vivo, a actividade antibiótica dos carbapenems e as cefalosforinas Antimicrob. Agents Chemother. 2017, 61, e01172; J. Antimicrob. Chemother. 2016, 7, 2171; Antimicrob. Agents Chemother. 2019, 63, e01092-19). Tamén estamos a estudar a evolución in vivo da resistencia bacteriana, como fonte de inspiración no deseño de fármacos (J. Antimicrob. Chemother. 2020, 75, 3209-3217; J. Antimicrob. Chemother. 2021, 76, 91-100).
AXENTES SILENCIADORES – A atenuación da virulencia bacteriana (Quorum-Quenching) fai que a bacteria perda a súa capacidade de causar a infección, e en consecuencia pode ser eliminada polo sistema inmune do hóspede. Centrámonos en desactivar o regulador transcripcional LasR de detección do quórum (QS), que é o regulador mestre de QS en P. aeruginosa, que modula a expresión de máis de 300 xenes (ACS Nano 2015, 9, 5567). Tamén estamos interesados no deseño de inhibidores intelixentes do enzima dehidroquinasa tipo I (DHQ1), unha diana bacteriana anti-virulencia, que actúa como un factor de virulencia in vivo. Temos desenvolvido diversos compostos funcionalizados con electrófilos pouco reactivos, capaces de modificar selectivamente residuos de lisina (Org. Chem. Front. 2019, 6, 3127), e demostramos que DHQ1 cataliza o seu autoinmolación transformando un grupo 2‐hidroxietilamonio nun epóxido que desencadea a inhibición irreversible soamente cando se unen á súa diana (J. Am. Chem. Soc. 2015, 137, 9333; Chem. Eur. J. 2020, 26, 8035).
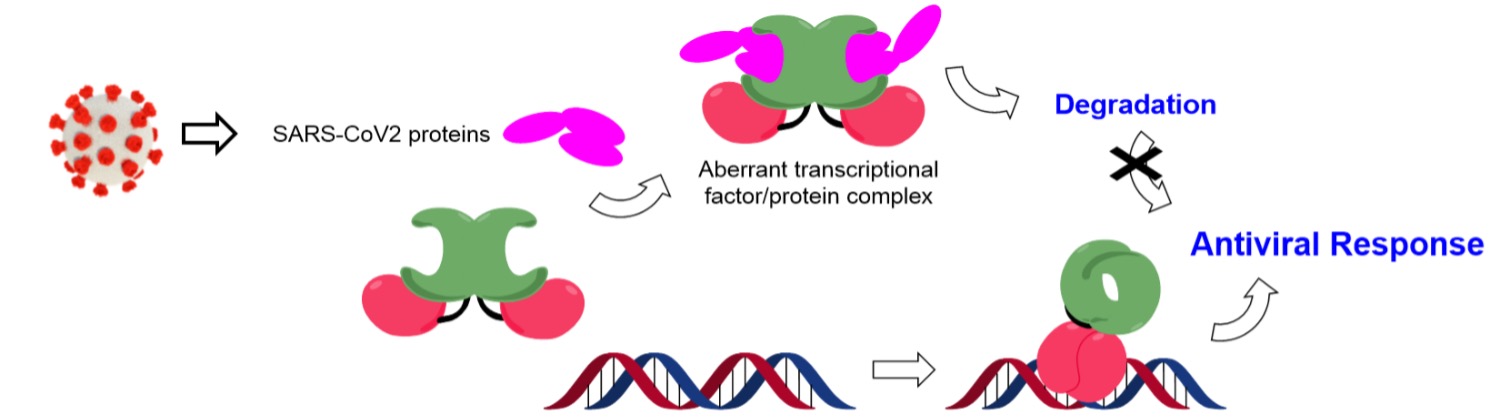
Aproximacións para Cancelar os Mecanismos de Evasión e Supresión Inmune do SARS- CoV-2
A pandemia da enfermidade COVID-19 causada pola infección polo novo coronavirus 2019 (SARS- CoV-2) converteuse nunha emerxencia de saúde pública mundial, cuxo estudo tamén é o foco da nosa investigación. A infección por SARS-CoV-2 provoca o trastorno da inmunidade natural e adaptativa que provoca dano tisular e inflamación sistémica, que é a principal causa de morte dos pacientes con esta enfermidade. As proteínas virales a miúdo xogan un papel fundamental ao interferir coa resposta inmune do hóspede, con todo, actualmente descoñécese o mecanismo molecular que subxace á modulación das vías de sinalización inmune polo SARS-CoV-2. Estamos interesados en explorar o mecanismo molecular detrás deste complexo e desafiante proceso viral, así como no desenvolvemento de pequenas entidades químicas capaces de modularlo. Estamos centrados no estudo en detalle atómico do mecanismo de activación da vía de sinalización do interferón que establece o estado antiviral, que parece estar modulado pola expresión de pequenas proteínas capaces de inactivar a produción de interferón nas primeiras etapas da infección. De feito, a eliminación sistemática das funcións moduladoras de interferón do virus é un enfoque moi prometedor para o desenvolvemento de vacinas.
Ferramentas para o Deseño
O noso grupo de investigación especializouse na aplicación de poderosas ferramentas complementarias no deseño e o descubrimento de fármacos, como a cristalografía de raios-X de proteínas, e os estudos computacionais de alto nivel que teñen en conta o dinamismo e o mecanismo de acción da biomacromolécula, simulacións de Dinámica Molecular (DM), estudos de mecánica cuántica/mecánica molecular (QM/MM). Isto permítenos abordar proxectos máis complexos desde unha perspectiva multidisciplinar máis aló da química, así como coñecer en profundidade os factores crave para a interacción, a inhibición ou a modulación da actividade por parte do ligando. Temos resolto máis de 30 estruturas cristalográficas de diversos complexos enzima/ligando e aductos, como son os obtidos coa siquimato quinasa procedente de M. tuberculosis, a deshidroquinasa tipo II procedente de M. tuberculosis e H. pylori ou a deshidroquinasa tipo I procedente de S typhi e S. aureus. Tendo en conta o movemento intrinseco do enzima, desenvolvemos tamén potentes inhibidores competitivos reversibles con boa actividade in vitro e in vivo fronte a H. pylori (Chem. Eur. J. 2016, 22, 17988). Ademais, empregando simulacións QM/MM puidemos determinar o mecanismo catalítico da deshidroquinasa de tipo II (DHQ2), unha recoñecida diana terapéutica para o descubrimento de antibióticos, identificar os aspectos determinantes das diferenzas de actividade catalítica entre enzimas homólogas (Org. Biomol. Chem. 2018, 16, 4443), así como o mecanismo de modificación covalente selectiva de enzimas/proteínas, ben inducido polo propio ligando (Chem. Eur. J. 2020, 26, 8035; Org. Chem. Front. 2019, 6, 3127), ou pola xeración de especies reactivas tras irradiación (J. Org. Chem. 2018, 83, 13019; Chem. Sci. 2017, 8, 2621; Org. Chem. Front. 2019, 6, 99; Chem. Eur. J. 2020, 26, 15922;).




